
Abbildung 1 Titelbild Blog #56 Stefan Moser
DoE Blog #057 – Von Pflanzen, Peaks und Plausibilität: Korrelation trifft Kausalität
Was macht ein Thema wie Non-Target Screening (NTS) in einem DoE-Blog?

Abbildung 2:Starter Bild der Presentation auf der ICNTS-Konferenz’25 in Erding
Im Rahmen der diesjährigen ICNTS-Konferenz hatte ich die Gelegenheit, einen Vortrag zu halten – entstanden aus einem gemeinsamen Projekt mit AFIN-TS. Ein Format, das mir erlaubt hat, tiefer in die Welt des Non-Target Screenings (NTS) einzutauchen und die Verbindung von analytischer Chemie, Multivariater Statistik (MVDA) und Design of Experiments (DoE) greifbar zu machen.
Fachlich anspruchsvoll, Publikum hochspezialisiert – und gerade deshalb spannend, das Ganze hier einmal für eine breitere DoE-Praxiswelt aufzubereiten.“
In diesem Blogbeitrag möchte ich nun die Essenz dieses Vortrags aufbereiten – angepasst für meine Leserinnen und Leser, die oft aus ganz unterschiedlichen Branchen kommen und sehr verschiedene Erfahrungen mit DoE mitbringen.
Wie immer gilt: Keine trockene Statistik, kein PowerPoint-Showdown – sondern praxisnahes Erklären mit Bodenhaftung.

Was Pflanzen mit Statistik zu tun haben
Unser Modell: Phragmites australis, das Gewöhnliche Schilfrohr. Nicht unbedingt das Erste, woran man bei High-End-Analytik denkt, oder?
Doch genau dieser biologische „Datenlogger“ hilft uns, Spurenstoffe wie Medikamente aus der Umwelt aufzuspüren. Pflanzen in pflanzenbasierten Klärsystemen nehmen Substanzen auf – und speichern Informationen über Umweltbelastung in ihren Zellen.
Die eigentliche Herausforderung: Wie übersetzen wir pflanzliche Zellinformationen in messbare Signale?“
In unseren Versuchsreihen setzen wir auf kontrollierte Bedingungen im Labor: Pflanzen wie Phragmites australis oder Lemna minor dienen dabei nicht nur als Umweltindikatoren, sondern als Modellorganismen. Unter exakt einstellbaren Bedingungen – von der Expositionsdauer bis zur Nährlösung – simulieren wir reale Umweltprozesse im Miniaturformat.
Diese Labormodelle liefern uns nicht nur chemische Fingerabdrücke, sondern erlauben auch gezielte Hypothesentests:
- Wie verändern sich Metabolitenprofile bei steigenden Konzentrationen eines Wirkstoffs?
- Wie wirken sich unterschiedliche Pflanzenteile oder Lösungsmittel auf die Nachweisbarkeit aus?
Genau hier entsteht die Brücke zwischen Biologie, Chemie – und Statistik.
Die Antwort ist ein klar strukturierter Ablauf – und der beginnt nicht bei der Statistik, sondern bei der Planung.
Von der Substanz zur Struktur: Ein analytischer Roadtrip
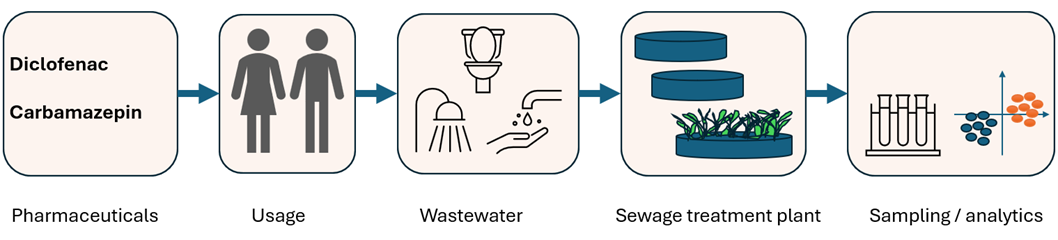
Abbildung 3: Flowchart von der Medikamenten Einnahme bis zur (in)-direkten Detektion in der Natur
Ein kurzer Überblick, wie Medikamente in die Umwelt geraten: Einnahme, Ausscheidung, Dusche, Waschmaschine, Abwasser. Viele Wirkstoffe überstehen die Kläranlage – und landen in Pflanzen, Boden oder Wasser.
Was dann passiert, zeigt eine Animation aus meiner Präsentation:

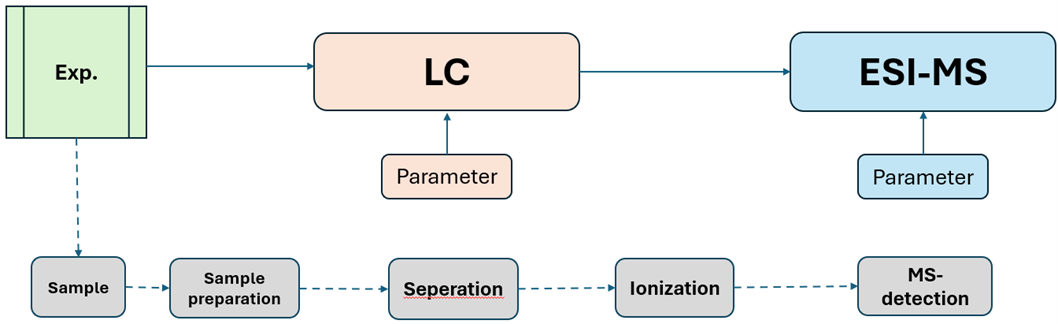
Bildbeschreibung: Ein animiertes GIF zeigt die Schritte: Probenaufbereitung → Trennung (HPLC) → Ionisierung (ESI) → Massendetektion (MS).
In der Praxis bedeutet das: Wir erzeugen riesige Datensätze. Tausende Signale pro Probe. Jede Probe ein chemischer Fingerabdruck.
Diese Rohdaten erklären sich leider nicht von selbst – Auch hilft hier in den sehr großen Tabellen auch keine einfache Einfärbung, um Muster erkennen zu können.
Struktur statt Datendschungel: Datenbank & Vorbereitung
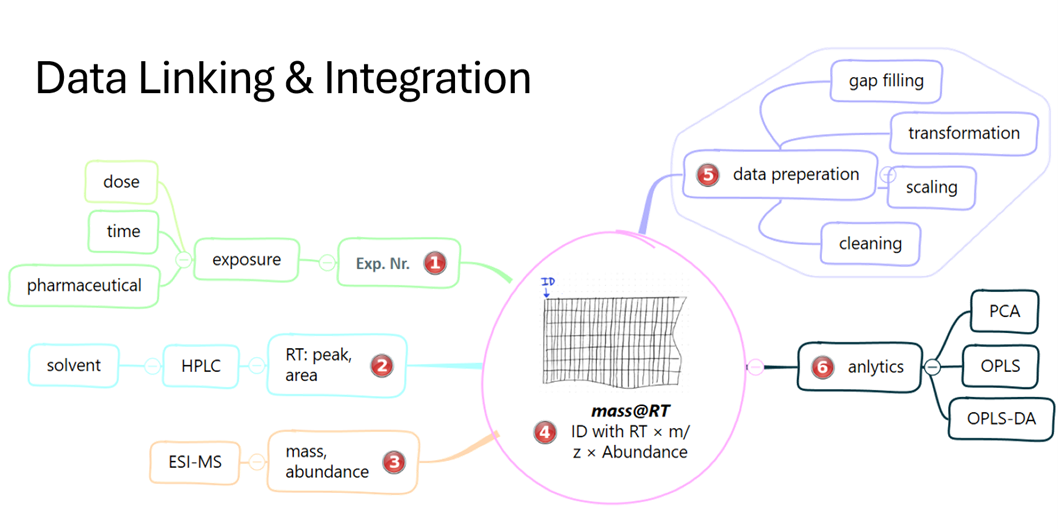
Zuerst werden die Massendaten bereinigt: Addukte, Isotope, Lücken, alles wird in Tools wie mzMine aufgeräumt.
Dann wandern die Signale in eine Datenbank – verbunden durch Retentionszeit, m/z-Wert und Signalstärke.
Erst hier entsteht überhaupt die Basis, um analytisch oder statistisch weiterzudenken.
Doch hier kommt ein Begriff ins Spiel, der für manche zunächst abstrakt wirkt – in Wirklichkeit aber Ordnung ins Datenchaos bringt:
PCA – Principal Component Analysis: Wenn Statistik Muster sichtbar macht

Bildbeschreibung: Das GIF erklärt, wie aus einem Datenblatt mit Messwerten durch Centering und Projektion in den Hauptkomponentenraum (PC1/PC2) ein 2D-Plot entsteht, der Cluster und Trends zeigt.
Bevor ein Signal statistisch verarbeitet wird, braucht es Vorbereitung: Skalierung, Transformation und die Behandlung fehlender Werte. Oft unterschätzt – aber entscheidend. Denn nur wenn wir biologisch relevante Unterschiede von technischem Rauschen trennen, entstehen robuste Modelle.
Wir arbeiten hier z. B. mit UV, Pareto-Skalierung und log-Transformation, um Ausreißer abzufedern und Unterschiede zwischen stark und schwach exprimierten Substanzen vergleichbar zu machen. Der Effekt? Deutlich stabilere PCA- und OPLS-DA-Modelle – und damit belastbarere Hypothesen.
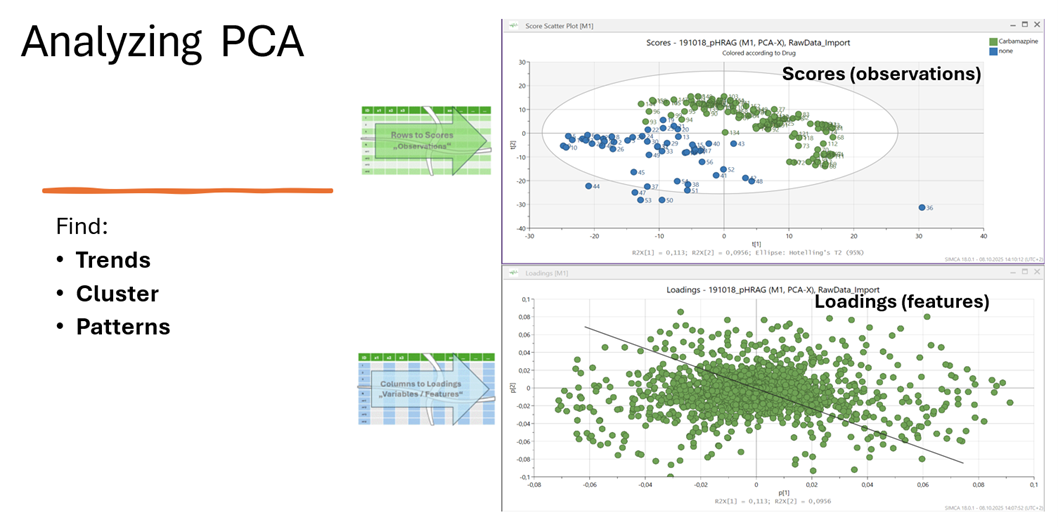
Die Principal Component Analysis (PCA) PCA lüftet den Statistikdschungel und zeigt, welche Bäume wirklich im Weg stehen – oder den Pfad markieren. Oder anders gesagt: Es zeigt, was unterscheidet sich am deutlichsten zwischen den Proben.
- Jede Messprobe wird ein Punkt (Score-Plot)
- Jede Substanz ein Richtungspfeil (Loading-Plot)
- Gruppen, Ausreißer, Trends? Plötzlich sichtbar
Aber: PCA zeigt Korrelation – nicht Kausalität. Und genau hier trennt sich Spielerei von echter wissenschaftlicher Erkenntnis..
Von der Projektion zur Deutung: Was Score- und Loadingplots verraten
Bevor wir zu der Frage kommen, warum Korrelation nicht gleich Ursache ist, lohnt sich ein genauer Blick auf die Methoden, mit denen wir überhaupt Muster erkennen:
OPLS-DA im Einsatz
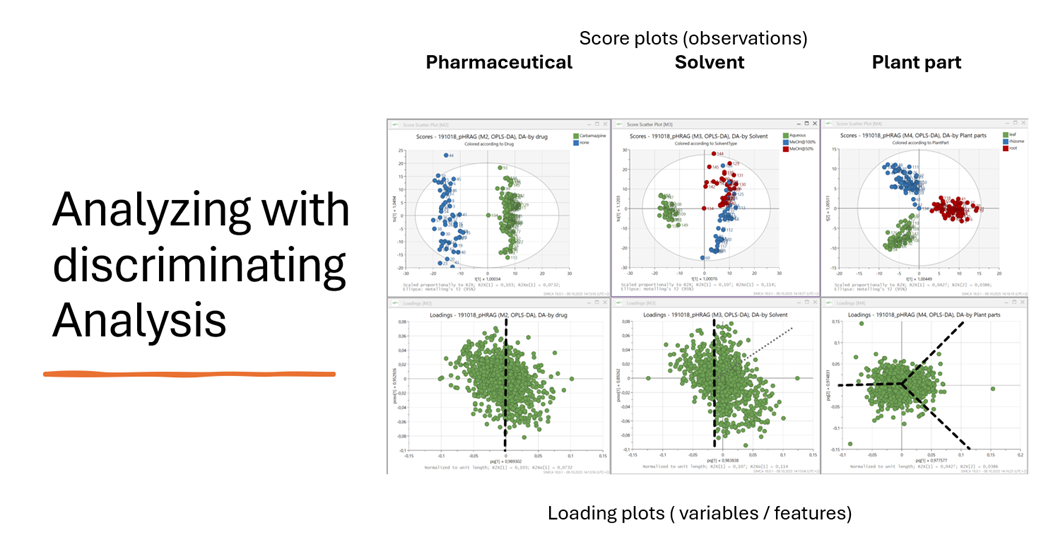
Nach der PCA kommen weitere Methoden ins Spiel: OPLS-DA (Orthogonale Partial Least Squares Diskriminanzanalyse) ist eine Weiterentwicklung, die gezielt zwischen zwei Gruppen trennt – zum Beispiel: behandelt vs. unbehandelt.
Im sogenannten Score-Plot sehen wir jede Probe als Punkt im Raum. Wenn sich Gruppen trennen, heißt das: Es gibt Unterschiede im chemischen Fingerabdruck. Der dazu gehörige Loading-Plot zeigt, welche Substanzen dafür verantwortlich sind.
Die Kombination dieser beiden Plots erlaubt Hypothesen wie:
- „Diese Pflanzen zeigen unter Diclofenac-Einfluss andere Metaboliten.“
Ein spannender Nebenaspekt, der gerne untergeht: Schon die Wahl des Lösungsmittels oder des untersuchten Pflanzenteils beeinflusst das metabolische Fingerprint. In unseren Tests zeigten Blätter, Stängel und Wurzeln völlig unterschiedliche Profile – ebenso wie Ethanol, Methanol oder Wasser als Extraktionsmittel.
Was zunächst wie ein Störfaktor wirkt, entpuppt sich als echter Erkenntnisgewinn: Denn genau diese Unterschiede helfen uns dabei, die Komplexität der Daten besser zu durchdringen und gezielter auszuwerten.
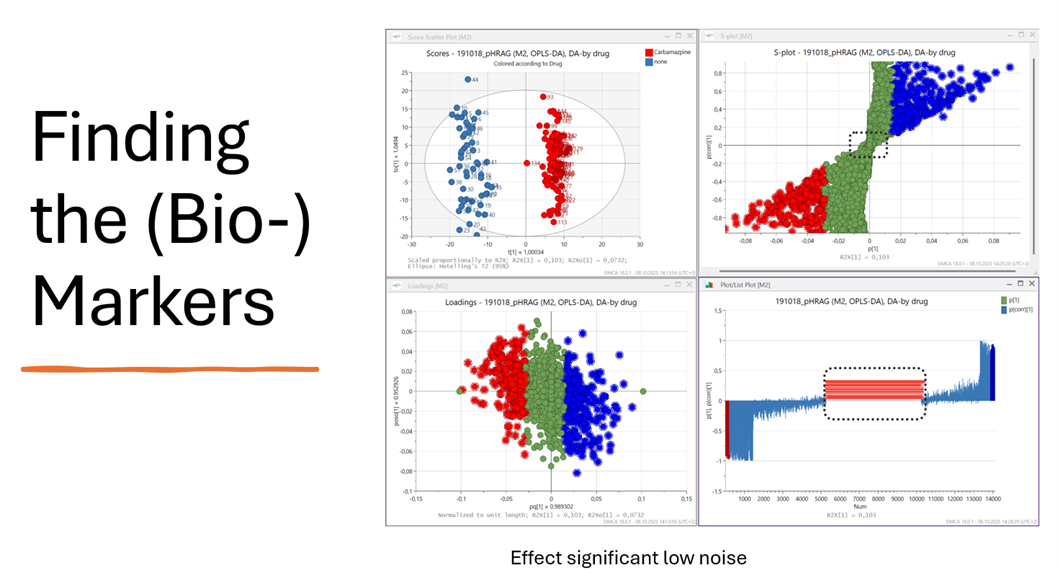
Abbildung 5: S-Plot aus einer OPLS-DA Analyse zur Schematischen Darstellung von Markern
Bildbeschreibung: Score- und Loading-Plots zeigen gruppenspezifische Unterschiede; das S-Plot färbt diese nach ihrer Wichtigkeit und trennt Signal von Rauschen. In unserem Beispiel triggert Diclofenac pflanzliche Abwehrreaktionen.
Noch klarer wird es mit dem S-Plot: Eine Darstellung, bei der Signifikanz und Relevanz gemeinsam gezeigt werden. Die weit ausliegenden Punkte (ganz links/rechts) sind besonders interessant: potenzielle Marker.
Natürlich freuen wir uns über „Marker-Substanzen“ wie ein auffälliges Quercetin-Signal. Aber die eigentliche Erkenntnis liegt im Muster: Welche Kombination von Substanzen verändert sich gemeinsam? Welche Cluster reagieren auf den Stressor?
Wir sprechen hier bewusst von systemischen Fingerprints – nicht von Einzelreaktionen. Und genau das ist der Kern moderner Umweltanalytik: Wir verstehen das biologische System als Ganzes.
All das ist faszinierend – aber es bleibt ein Blick auf Zusammenhänge. Erst der nächste Abschnitt zeigt, warum das allein nicht reicht.
Korrelation ist nicht Kausalität – und genau da liegt die Falle

Eine meiner Lieblingsfolien zeigt ein Baby, das von einem Storch gebracht wird. Statistisch nachweisbar! In bestimmten Regionen. Aber eben keine Kausalität.
Und tatsächlich Korreliert in Oldenburg die Geburtenrate mit der Storchen-Population.
Große Datenmengen sind wunderbar – aber sie machen es auch einfacher, sich zu täuschen:
- Nur weil etwas gemeinsam auftritt, ist es nicht verbunden
- Kein Nachweis ist nicht gleich kein Effekt
- Und auch bei Big Data: Verzerrung und Rauschen bleiben
Merksatz: „Nur weil das Dach eingedrückt ist, war es nicht automatisch die Katze. Genau so führt auch in der Statistik nicht jede Korrelation zur richtigen Ursache.
Oft wird DoE erst im Zusammenhang mit statistischer Auswertung erwähnt – dabei beginnt der methodische Vorteil deutlich früher: schon in der Planungsphase. Welche Konzentrationen, welche Pflanzengewebe, welches Lösungsmittel? Durch systematische Varianzplanung schaffen wir schon vor der ersten Messung eine belastbare Struktur.
Selbst das Setup der Messtechnik – vom Flussgradienten in der HPLC bis zur Ionisierungsquelle im MS – wurde gezielt mit DoE optimiert, um Rauschen zu reduzieren und Signale zu stabilisieren. Weniger Rauschen, mehr Reproduzierbarkeit, klarere Signale.
Warum DoE den Unterschied macht
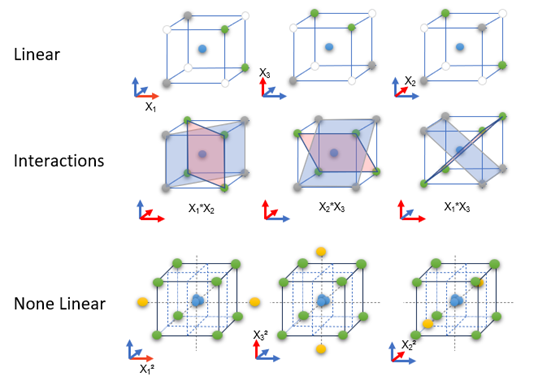
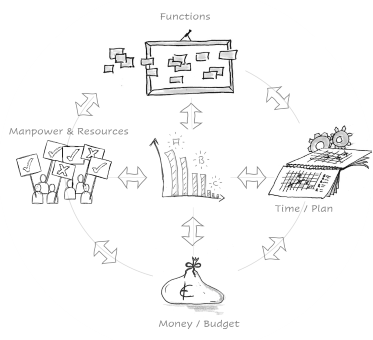
Abbildung 7: Typischer komplementärer „DoE Aufbau“ Screenen, Wechselwirkungen und Nichtlinearität. Rechts davon Schema zur Problemformulierung aus dem Projektmanagement
Genau an dieser Stelle kommt Design of Experiments (DoE) ins Spiel.
DoE ist Statistik mit Plan. Und der Unterschied ist gewaltig:
- Wir variieren mehrere Parameter gleichzeitig
- Wir können Interaktionen erkennen
- Wir vermeiden systematisch Zufallskorrelationen
- Wir schaffen Planbare Abarbeitungen
OFAT vs. DoE

Abbildung 8: Bart Simpson schreibt an die Tafel: „I will not use OFAT when DoE exists.“ Daneben die Nachteile: Kein Rauschentest, keine Wechselwirkungen, Überanpassung, ineffizient.
OFAT wirkt verlockend einfach – aber führt oft in die Irre: keine Wechselwirkungen, keine Robustheit, keine Sicherheit.
DoE dagegen erlaubt:
- Reproduzierbarkeit (Centerpoints!)
- Untersuchung nichtlinearer Effekte
- Globale Optimierung statt lokaler Bauchgefühl-Erkenntnis

Abbildung 9: Tabellarische Darstellung OFAT vers. DoE
Fazit: Von Mustern zur Mechanik
- Statistik zeigt Muster. DoE erklärt sie.
- Wenn wir wissen, wie Daten entstehen, können wir mehr aus ihnen machen als nur hübsche Plots.
- Wir erkennen systemische Zusammenhänge – und schaffen die Grundlage für sinnvolle Entscheidungen.
- DoE ist dabei kein Statistik-Add-on, sondern der rote Faden von Problemformulierung, über Versuche bis zur Handlungsempfehlung.
Aus einem pflanzlichen Fingerabdruck wird so ein methodisch abgesicherter Baustein für Umweltmonitoring und Prozessoptimierung.
Ausblick / Rückblick
👉 Vielleicht fragen Sie sich jetzt: Wo lauert in meinem aktuellen Projekt eine Zufallskorrelation? Oder: Welche Parameter plane ich eigentlich noch zu einseitig?
👉 Vielleicht gab’s einen Aha-Moment? Dann freue ich mich über ein kurzes Feedback – oder einen Kommentar, wo hat es bei Ihnen Klick gemacht.

Abbildung 10: Stefan Moser – Logo
Mehr aus Ihren Prozessen rausholen?
Ob DoE-Grundlagen oder Spezialthemen wie Troubleshooting, Screening, Optimierung, Mischungsdesigns oder Robustheit – ich unterstütze Sie mit praxisnahen DoE-Trainings, gezielter Beratung und methodischer Begleitung. Auch bei MVDA, DFSS und QFD bin ich an Ihrer Seite – vom ersten Workshop bis zur robusten Umsetzung.

